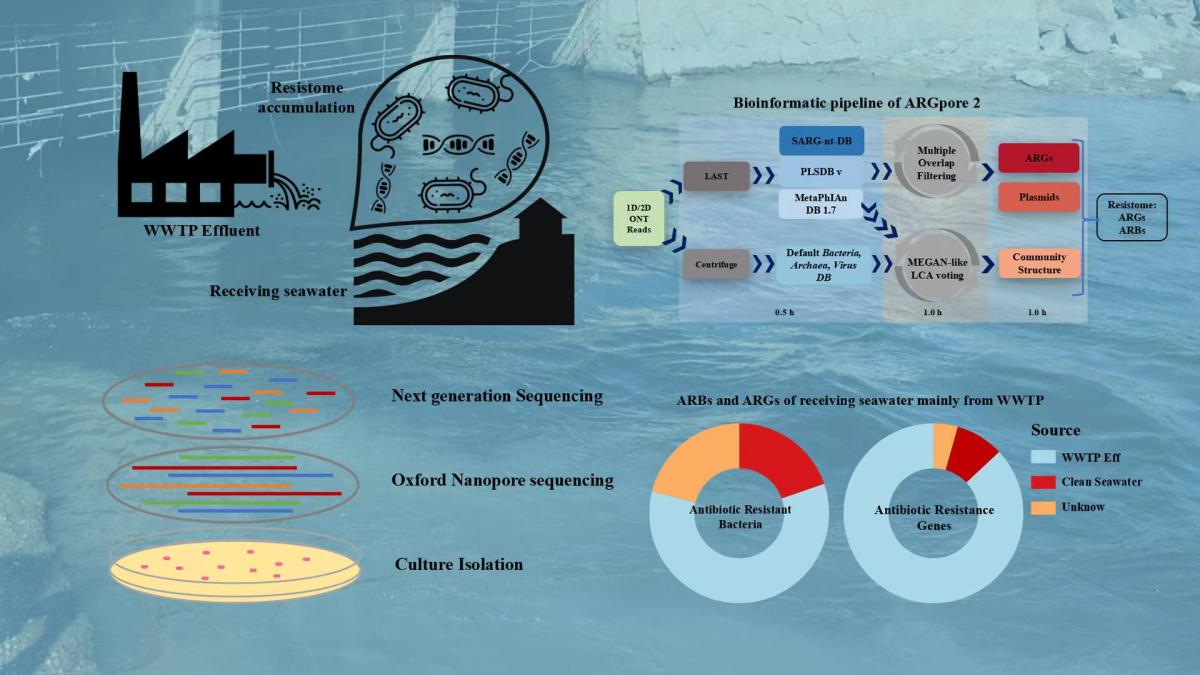
近日,南方科技大学环境科学与工程学院助理教授夏雨课题组在环境科学领域著名期刊 Water Research 发表了题为“基于纳米孔长读长宏基因组学揭示海边受纳水环境中耐药基因的富集机制”(Nanopore-based long-read metagenomics uncover the resistome intrusion by antibiotic resistant bacteria from treated wastewater in receiving water body)的研究论文。该论文利用第三代纳米孔测序技术(Nanopore Technology),对污水厂出水受纳水体中的耐药基因及其细菌宿主携带者的进行快速检测,并且自主开发了一个快速检测耐药基因和细菌宿主的宏基因组学分析工具ARGPore2(https://github.com/sustc-xylab/ARGpore2 ),实现了对耐药细菌及所携带耐药基因的精准检测。
 耐药基因已被世界卫生组织列为危害人类健康安全的亟待解决的污染物。目前由于抗生素在畜牧业、农业、医药业等的滥用,使得环境中的抗生素污染程度加剧,导致耐药性的微生物能够更好地适应含有抗生素的环境生存。污水厂用于处理居民生活、医院和工厂等排放的污染水体,是耐药基因传播和耐药性细菌富集的重要场所。因此揭示环境中耐药基因以及携带的细菌宿主和耐药基因在环境中的传播机制,这会为控制和阻断耐药基因肆意扩散危害人类健康安全的问题提供帮助。由此,为了更全面揭示耐药基因在受纳水体中传播的机理,本项工作将使用第三代纳米孔测序技术对受纳水体样品进行宏基因组测序和分析。目前第三代纳米孔测序技术能够在复杂样品中检测出菌群种类和耐药基因,但是并不能准确地鉴别出耐药基因的宿主。研究团队开发ARGPore2分析流程,结合第三代纳米孔测序数据,能够更准确地鉴定耐药基因以及定位耐药基因的携带菌种,从而为监测环境中耐药基因的传播提供了有效的手段。
耐药基因已被世界卫生组织列为危害人类健康安全的亟待解决的污染物。目前由于抗生素在畜牧业、农业、医药业等的滥用,使得环境中的抗生素污染程度加剧,导致耐药性的微生物能够更好地适应含有抗生素的环境生存。污水厂用于处理居民生活、医院和工厂等排放的污染水体,是耐药基因传播和耐药性细菌富集的重要场所。因此揭示环境中耐药基因以及携带的细菌宿主和耐药基因在环境中的传播机制,这会为控制和阻断耐药基因肆意扩散危害人类健康安全的问题提供帮助。由此,为了更全面揭示耐药基因在受纳水体中传播的机理,本项工作将使用第三代纳米孔测序技术对受纳水体样品进行宏基因组测序和分析。目前第三代纳米孔测序技术能够在复杂样品中检测出菌群种类和耐药基因,但是并不能准确地鉴别出耐药基因的宿主。研究团队开发ARGPore2分析流程,结合第三代纳米孔测序数据,能够更准确地鉴定耐药基因以及定位耐药基因的携带菌种,从而为监测环境中耐药基因的传播提供了有效的手段。
1. 鉴别耐药基因和耐药性细菌
ARGPore2是一款有效地鉴别耐药基因和耐药性细菌的分析工具。它使用LAST比对工具,对纳米孔测序得到的数据和整合耐药基因核酸数据库SARG进行比对,从而鉴别和计算出样品中检测的耐药基因的种类和丰度。研究团队人为制作了样品数据由从耐药基因数据库SARG抽取的序列和从KEGG数据库中抽取的非耐药基因的其他功能性基因的序列组成,去检测ARGPore2的鉴别耐药基因的准确性。通过检测,当使用参数中相似度的比例选择在75%以上时,能有95%以上的耐药基因被准确地识别和检测。同时,ARGPore2通过引用Centrifuge物种鉴别工具对样品中的菌群物种信息的分类结果,再使用LAST比对工具将分类到的细菌染色体的序列与MetaPhlan物种信息库进行比对,从而准确的鉴别出样品中的细菌种类和计算其在样品中的丰度。最后,ARGPore2通过结合从样品中检测出的耐药基因信息和菌种信息,以此准确定位出样品中的耐药性细菌。
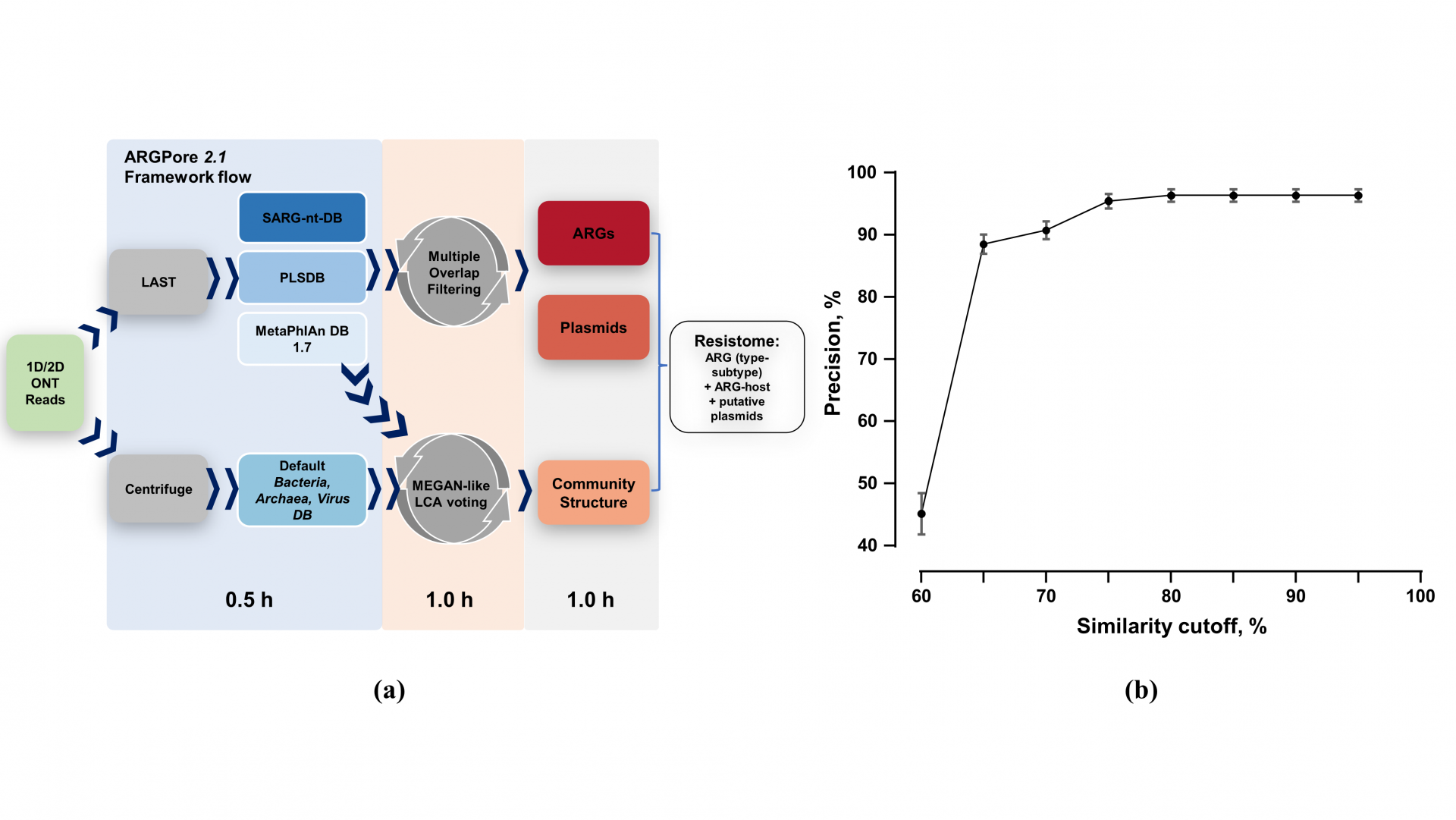
图 1 ARGPore2.0的分析流程和表现 (a) ARGPore2 的分析工作流程图解;(b) 在不同的相似度参数的选择下ARGpore2的准确性。
使用ARGPore2对3组纳米孔测序得到的数据分析得出,在受纳水体样品中,有380个耐药性细菌包含10种不同细菌种属被检测出来。相反,在Illumina测序对相同样品的测序结果分析得到,只有18个耐药性细菌从样品中检测得到。通过对比两种测序手段检测到的耐药性细菌的结果发现,纳米孔测序技术找到耐药性细菌中,大肠杆菌和克雷伯菌是主要的耐药基因携带者,这两类菌属也是环境中普遍携带耐药基因的主流菌属,而在Illumina测序序列组装的结果中却与此相反。
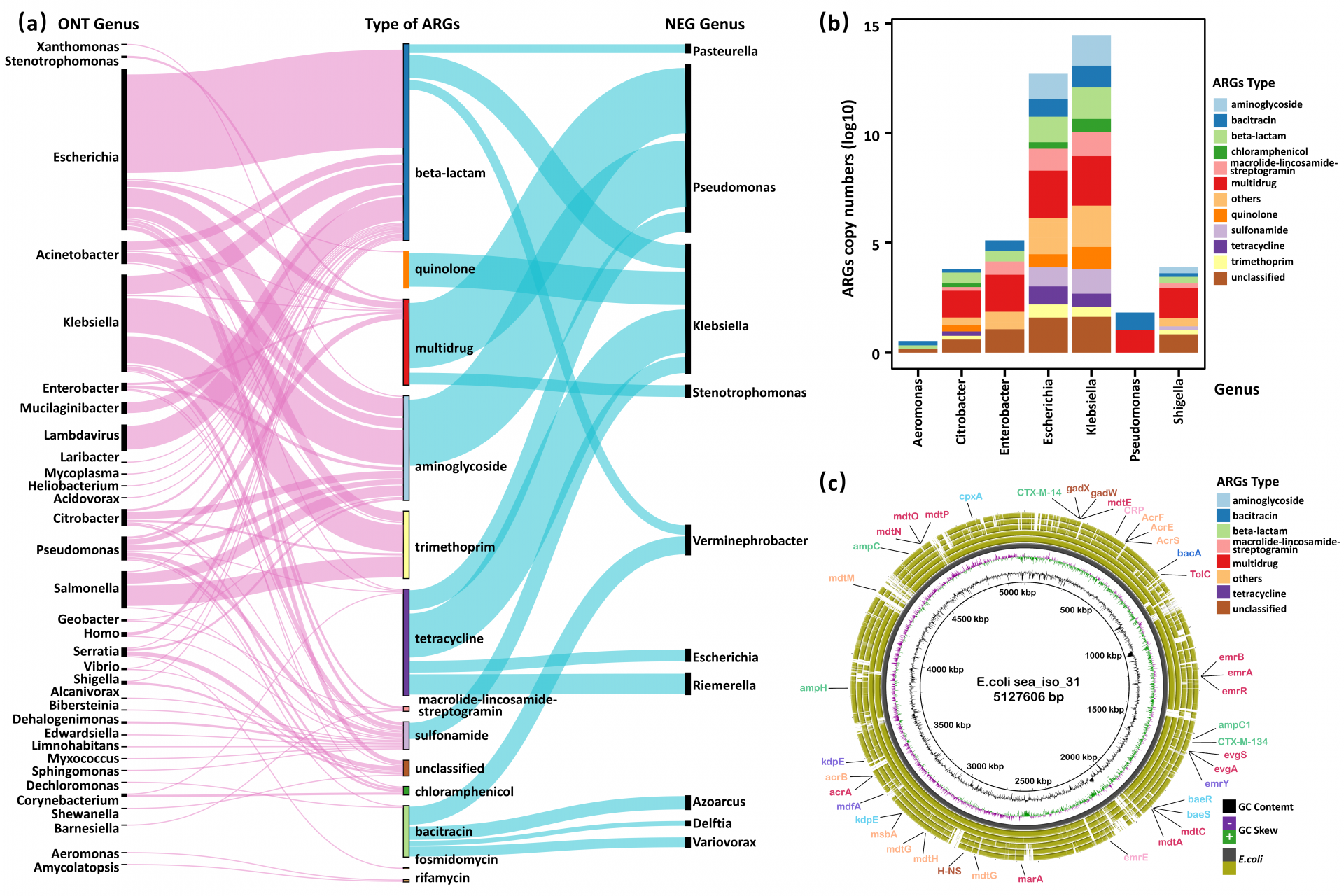
图 2比较纳米孔宏基因组测序和Illumin宏基因组测序序列组装分析耐药基因的结果。(a)在种属水平,比较纳米孔测序得到的耐药性细菌的检测结果和Illumina测序序列组转得到的耐药性细菌的检测结果。(b)24株从样品中随机分离培养的携带β内酰胺类耐药基因的细菌的耐药基因结果图谱。(c)BRIG基因图谱比较6株携带耐药性基因的大肠杆菌的染色体基因结构。
2. 耐药基因在污水厂接纳水体中富集
ARGPore2分析三代纳米孔宏基因组测序的样品结果得出,总共有65种耐药基因亚型和14种耐药基因大类被检测出来,其中与耐药基因库匹配的序列中识别出780个耐药基因,其中74%的耐药基因是定位在细菌基因组上。检测到受纳水体样品的耐药基因的平均浓度是对照海水样品的耐药基因的平均浓度的十倍,其中TEM-group1, OXA-group2, aac(6’)-I, aadA 和 dfrA7/A17等这类耐药基因呈现明显的上升趋势。其中,b内酰胺类耐药基因是受纳水体中的主要耐药基因类型,同时TEM-group的耐药性基因与医疗使用的抗生素的使用有密切的相关性。
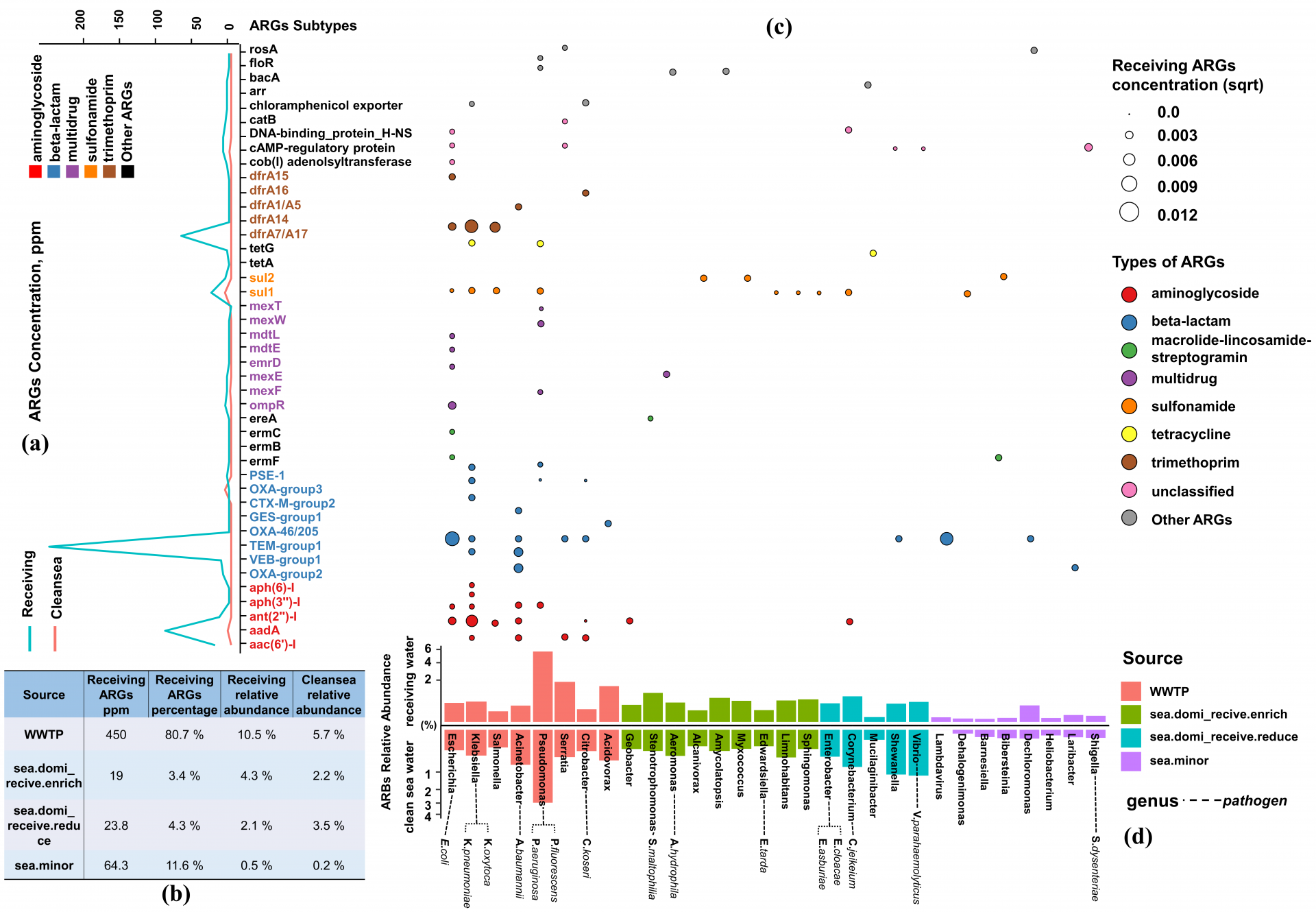 图 3受纳水体和对照海水的耐药基因型图谱。(a)比较受纳水体和对照海水中的耐药基因的平均浓度;(b)样品中耐药基因的细菌及特定的耐药性致病菌的相对丰度信息;(c)受纳水体中耐药基因的浓度分布;(d)耐药基因在各个样品中的相对丰度分布信息。
图 3受纳水体和对照海水的耐药基因型图谱。(a)比较受纳水体和对照海水中的耐药基因的平均浓度;(b)样品中耐药基因的细菌及特定的耐药性致病菌的相对丰度信息;(c)受纳水体中耐药基因的浓度分布;(d)耐药基因在各个样品中的相对丰度分布信息。
3. 耐药基因在接纳水菌群中的传播
通过对接纳水体样品、对照海水样品和污水厂出水样品的数据进行贝叶森溯源分析的结果得出,受纳水体样品中有85%的耐药基因是受到污水厂出水影响的,同时受纳水体中有70%的菌种受到污水厂出水的影响。在受纳水体样品中检测出187种潜在的致病菌菌种,其中98个携带耐药性致病菌的被鉴别出来。其中,肠杆菌属细菌,肺炎克雷伯菌,鲍曼不动杆菌和铜绿假单胞菌,这四种医院内感染高度耐药细菌也在受纳水体样品中被检测出来。
4. 受纳水体中可移动耐药基因的分布
在受纳水体和对照海水样品检测出的耐药基因结果发现,有74%的耐药基因是在细菌染色体上携带的。对比两类水体样品的耐药基因结果发现,抗膦胺霉素基因、抗氯霉素基因、抗喹诺酮基因、抗四环素基因等七种耐药基因只在受纳水体中检测得到。同时,受纳水体中的耐药基因有54%是由细菌的染色体所携带的。
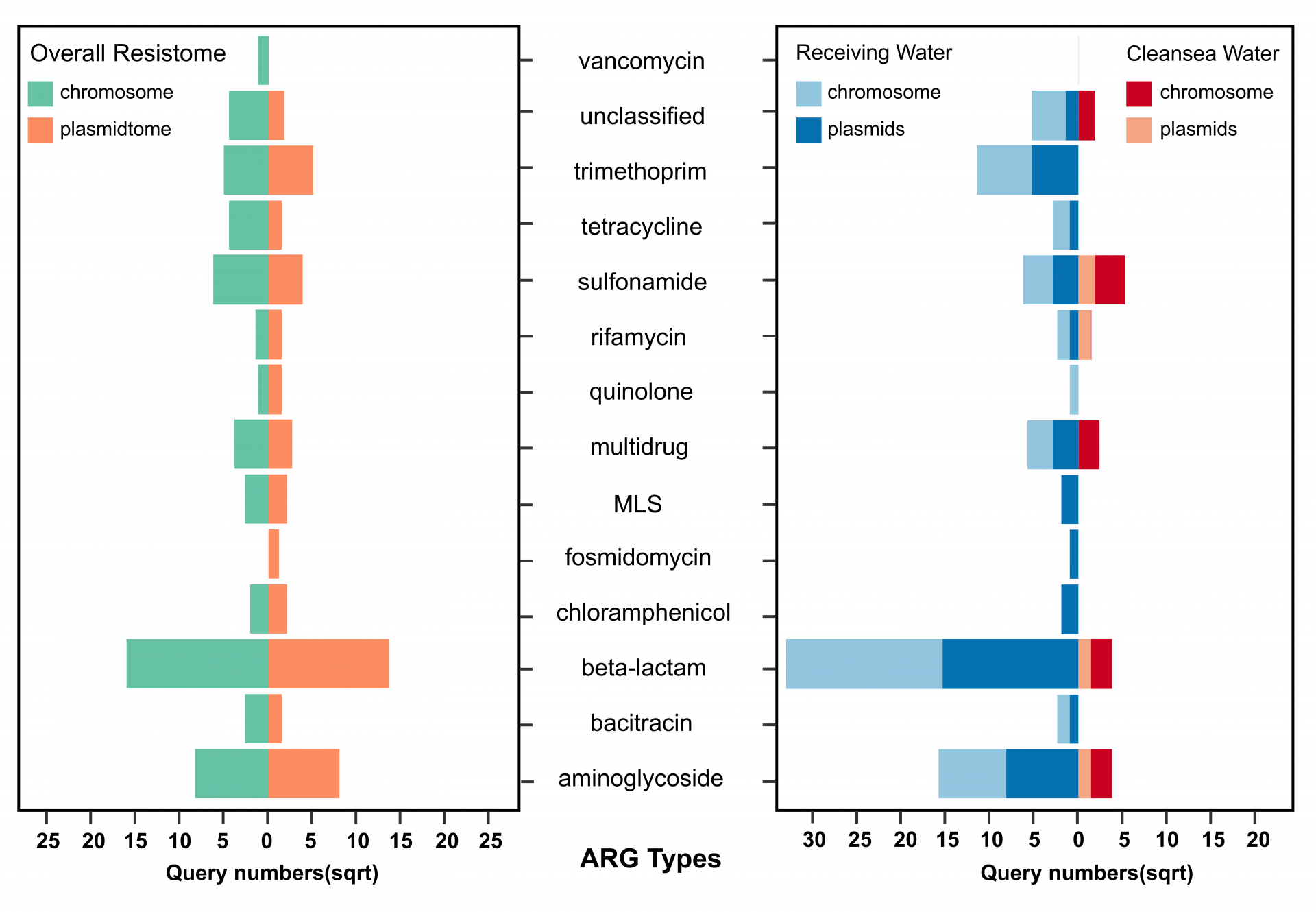
图 4左图为耐药基因在在细菌染色体和质粒上总体分布情况;右图为耐药基因分别在受纳水体和对照海水中的细菌的染色体和质粒上的分布情况。
通过对样品中的耐药基因在基因组上的分布发现,在含有转座子、整合子和重组酶的基因序列上,定位到16种耐药基因分别与这类可移动原件共存。这类可移动原件能够在样品菌群中发生水平基因转移,从而促进了耐药基因在样品菌群中的传播和转移。

图 5基因组上的可移动原件和耐药基因的分布图谱
本项工作应用纳米孔测序技术,结合自主开发的ARGpore2分析流程,准确地鉴别出环境样品中耐药基因和耐药性细菌,从而揭示海边受纳水体环境样品中耐药基因的富集原理。研究团队的结果表明,受纳水体中的耐药基因和菌群种类主要受到污水厂出水的影响,其中质粒和1类整合子是耐药基因水平转移的主要载体。本项目提供的耐药基因和耐药性细菌的鉴别和溯源方法,能过为监测和控制环境中耐药基因污染问题提供一个新的解决思路。
南方科技大学和哥本哈根大学联合培养博士生吴子麒为论文的第一作者,夏雨为通讯作者。南科大是论文第一单位。合作者包括香港大学教授张彤,香港大学博士车有等。该成果得到了国家自然基金和国家重点研发计划基金和南方科技大学启动基金的支持。
论文链接:https://doi.org/10.1016/j.watres.2022.119282
供稿:环境科学与工程学院
通讯员:晏梓添
主图:丘妍
编辑:朱增光




