近日,南方科技大学生命科学学院副教授翟继先团队结合三代测序技术,开发出一套能同时检测PolyA信息,Pol II转录位置和多内含子剪接情况的高通量全长单分子测序技术(图1)。相关技术已成功用于拟南芥转录及剪接相关研究,并以“FLEP-seq: simultaneous detection of RNA polymerase II position, splicing status, polyadenylation site, and poly(A) tail length at genome-wide scale by single-molecule nascent RNA sequencing”为题发表在国际知名方法学领域期刊Nature Protocols。
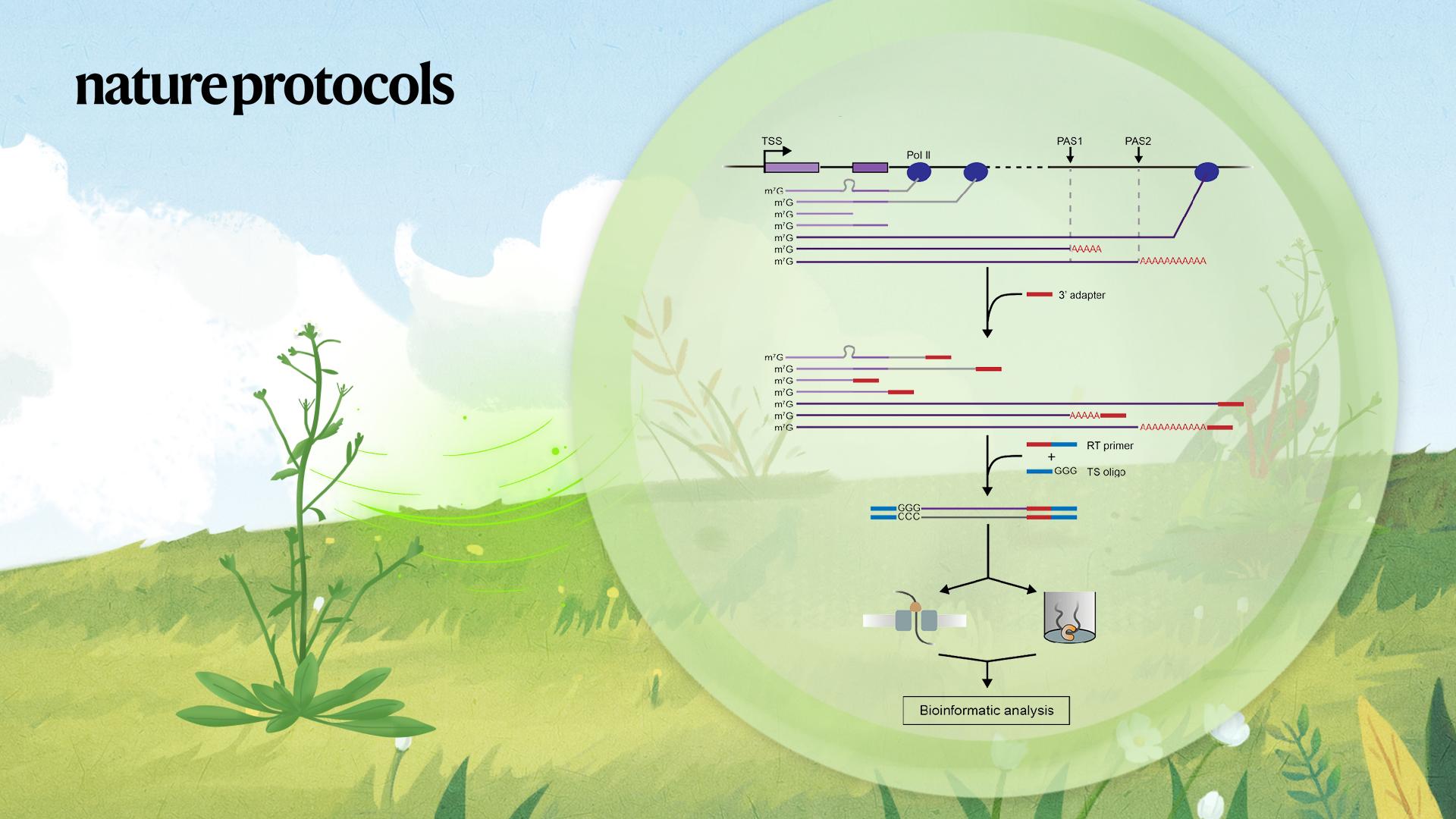
Pre-mRNA的加工和成熟是真核生物基因表达调控的重要步骤。从转录合成开始,至输出细胞核并在细胞质中翻译之前,pre-mRNA会通过一系列复杂的分子反应,包括5'端帽子的添加,内含子的剪切和3'端末端的多聚腺苷酸化。在真核生物中,大多数内含子都以共转录剪切的方式进行,在转录完成之前就已被去除。目前,剪切因子的招募以及剪接体的结构和组装过程已解析得比较清楚,但剪切动力学、共转录剪切的具体进程和3'末端加工过程仍有待进一步研究。
近年来,随着测序技术的发展,以Nanopore和PacBio为代表的三代测序技术逐渐成熟。二者产出的平均测序读长在kb级别,甚至高达几十kb,相比Illumina等二代测序平台bp级别的读长,优势明显。三代文库构建时无需将基因组/转录本打断,信息分析无需对测序片段进行组装,即可精准重构基因组和转录组全貌。目前在高重复和高杂合基因组组装、可变剪切位点鉴定、融合基因和大片段结构变异等研究中得到了越来越多的应用。将三代测序技术运用于转录加工过程的研究,不仅可区分每种类型的转录异构体,还能直接对每一条新生RNA上的每个内含子在转录过程中的剪接进程、转录终止过程中的polyA加尾和末端加工过程进行观察。
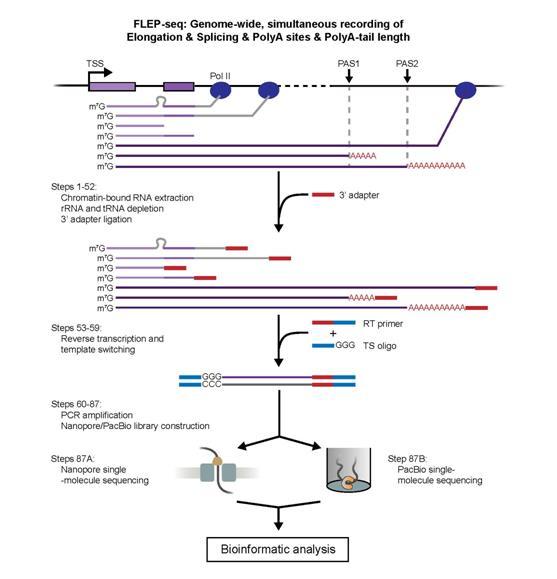
图1. FLEP-seq流程示意图
研究团队开发的方法能捕获包括Pol-II延伸过程中的转录本、已转录过polyA位点的read through转录本、以及已完成或正在进行加A的转录本在内的多种RNA转录、剪切和加工中间体,并同时对每个RNA分子上的内含子剪切状态、Pol-II转录位置、polyA位置以及polyA长度等信息进行检测(图2)。这为研究人员追踪并研究植物mRNA转录、剪切和加工的动态过程提供了重要的新工具。
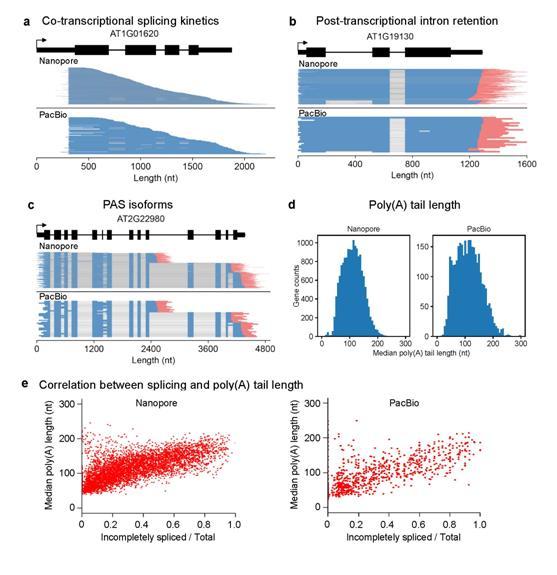
图2. FLEP-seq应用示例
课题组研究助理教授龙艳萍、贾津布为该论文共同第一作者,翟继先为该论文的通讯作者,南科大是论文第一单位。该研究得到了科技部国家重点研发计划、广东省创新创业团队、深圳市科创委以及广东省植物细胞工厂分子设计重点实验室基金的资助。
论文链接:https://www.nature.com/articles/s41596-021-00581-7
供稿:生命科学学院
主图:丘妍
通讯员:付文卿
编辑:朱增光




